2022年10月27日,天津医科大学基础医学院生化系张恒团队联合中国科学院武汉病毒所邓增钦团队,在Nature Microbiology期刊发表了题为Structure and function of a bacterial type III-E CRISPR–Cas7-11 complex的研究论文。该研究通过解析Cas7-11复合物不同状态的冷冻电镜结构,利用大量生化实验阐明了Cas7-11加工前体CRISPR RNA(pre-crRNA),识别和切割targetRNA的机制。研究还发现Cas7-11识别target RNA后,能够引起Csx29的构象变化,很可能激活其蛋白酶活性发挥免疫功能。
CRISPR-Cas系统是细菌和古菌抵抗可移动遗传原件入侵的适应性免疫系统,可以将其分为两类,第1类:由多个效应蛋白组成的蛋白复合物行使功能,包括I,III和IV型;第2类由单个效应蛋白行使功能,包括II,V和VI型。最近研究人员鉴定了一种新型III-E亚型CRISPR-Cas系统(ref 1)。该系统在哺乳动物细胞进行RNA打靶时具有较高的活性及较低的毒性(ref 2)。不同于之前发现的多亚基III型系统,它由4个Cas7和1个Cas11结构域融合形成一个大的效应蛋白Cas7-11/gRAMP,在crRNA指引下对靶标RNA进行切割。Cas7-11能与一种Caspase-like蛋白酶(Csx29/ TPR-CHAT)相互作用(ref 3),形成由CRISPR引导的Caspase复合物(Craspase),因此该系统可能同时兼具核酸酶与蛋白酶活性,暗示着一种新型抵抗噬菌体侵染的机制。
作者首先借助冷冻电镜解析了Candidatus ‘Scalindua brodae’菌株的Cas7-11-crRNA(SbCas7-11-crRNA)二元复合物结构,发现Cas7-11由四个结构相似的Cas7以及一个Cas11和一个IPD结构域组成(图1)。IPD是一段位于Cas7.4中间的插入序列,目前对其结构与功能的研究尚不清楚。然而作者将IPD删除之后,并未影响targetRNA的识别和切割,提示可以通过将IPD删除构建小型化的基因编辑工具。
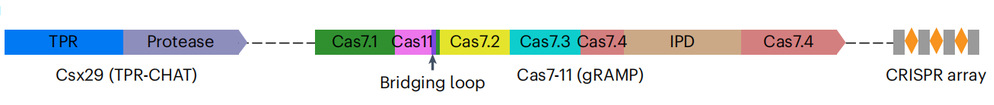
图1 III-E型CRISPR-Cas系统的基因结构
在传统的III型CRISPR-Cas系统中,Cas6蛋白负责pre-crRNA加工成熟,而Cas7-11蛋白本身就具有相应的pre-crRNA加工能力。Cas7-11–crRNA结构显示成熟后的crRNA的重复序列结合在Cas7.1结构域,因此推测Cas7.1结构域可能具有加工pre-crRNA的能力。接下来作者设计了一系列突变实验来寻找Cas7.1的活性位点。由于单体SbCas7-11不稳定,作者选用来自Desulfonema ishimotonii物种的同源蛋白DiCas7-11进行实验。结果表明DiCas7-11的4个突变体(W20A、R26A、H43A和Y55A)都能够明显阻碍pre-crRNA的加工(图2),证明了Cas7.1结构域在pre-crRNA切割中的重要作用。其中W20和R26在Cas7-11同源物中严格保守,但H43被SbCas7-11中的苏氨酸残基(T45)取代,Y55被苯丙氨酸残基(F57)取代。

图2野生型(WT)和突变型DiCas7-11蛋白的体外pre-crRNA切割实验。括号中为SbCas7-11对应残基(绿色)。
Cas7-11有两个targetRNA切割位点,间隔6个核苷酸,称为位点1和位点2。Cas7-11-crRNA-target RNA三元复合物结构发现crRNA序列中的两个核苷酸,4A和10U,分别被Cas7.2和Cas7.3结构域的b-指结构翻转,提示targetRNA切割很可能发生在这两个位点附近。在III-A/B系统中,Csm3/Cmr4亚基通过催化loop中的一个保守天冬氨酸残基发挥RNase活性。作者通过对比SbCas7-11和Csm复合物的结构,发现在Cas7.3中存在一个对应的保守的天冬氨酸残基(D698)。与预期的一样,D698A突变几乎消除了2位点的targetRNA切割(图3左)。然而,在Cas7.2中的对应位置包含一个非保守的丝氨酸残基(S457),并且S457A突变对1位点的切割影响不大。有趣的是,在同一催化口袋中的酸性残基D547的突变,几乎消除了SbCas7-11在1位点的切割活性(图3中)。更有趣的是,序列比对显示,在大多数Cas7-11同源物中,这两个相应位置上一般只会出现一个酸性氨基酸。例如,SbCas7-11有S457-D547,而DiCas7-11在对应的位置上是D429-N518。因此,作者提出了在这两个位置上的氨基酸可能存在功能冗余的假设,并做了功能丧失和功能获得突变来验证这一假设。与预期的一样,结果表明Cas7.2催化口袋的这两个活性位点酸性残基可能在功能上是等价的:两个位置的任何一个天冬氨酸残基都可以对targetRNA进行切割(图3右)。
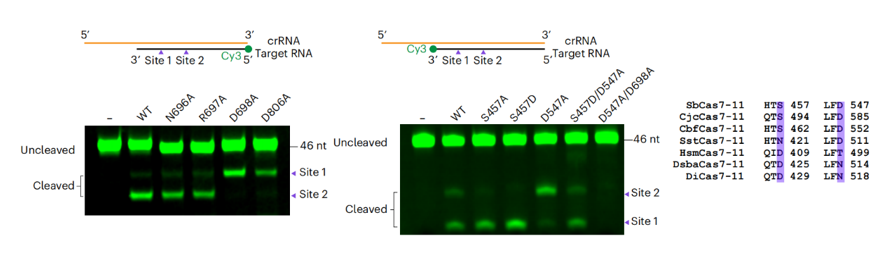
图3左,Cas7.3结构域突变的体外target RNA切割。中,Cas7.2结构域突变的体外target RNA切割。
右,Cas7-11同源物的序列比对。
为了研究Cas7-11如何调控Csx29的机制,作者解析了Cas7-11-crRNA-Csx29三元复合物和Cas7-11-crRNA-target RNA-Csx29四元复合物的电镜结构。结构揭示Csx29在targetRNA的存在下发生了明显的构象变化:TPR区域靠近Cas7-11,而蛋白酶PS结构域远离Cas7-11,使得Csx29的两个催化残基H585和C627在空间上更加靠近,暴露出其催化口袋(图4)。因此,target RNA的结合很可能可以激发Csx29蛋白酶活性,从而与Cas7-11协同抵抗外源核酸的入侵。该团队关于Csx29蛋白酶活性的测定及切割底物的鉴定等后续工作也证实了这一推断(该部分研究工作正在underrevision)。
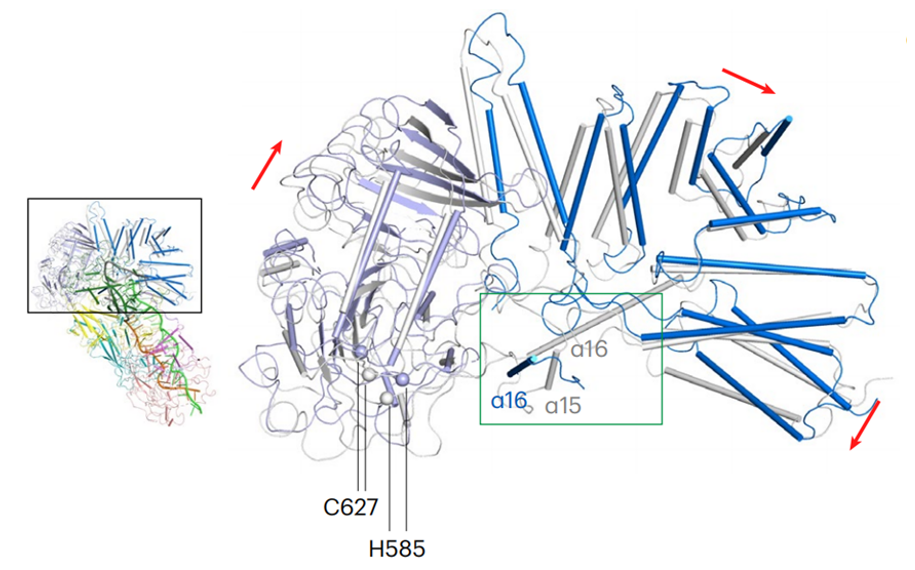
图4Csx29未结合target RNA和结合target RNA时的构象变化
该研究阐述了Cas7-11加工成熟pre-crRNA和切割targetRNA的机制,以及Cas7-11对Csx29活性的调控机制(图5)。该研究极大地促进了人们对于CRISPR系统的理解,并为Cas7-11作为安全高效靶向RNA编辑工具的工程化改造提供了结构基础。同时该系统所具有的RNA指导的蛋白酶活性可能为生命科学研究带来新的视角和新的工具。
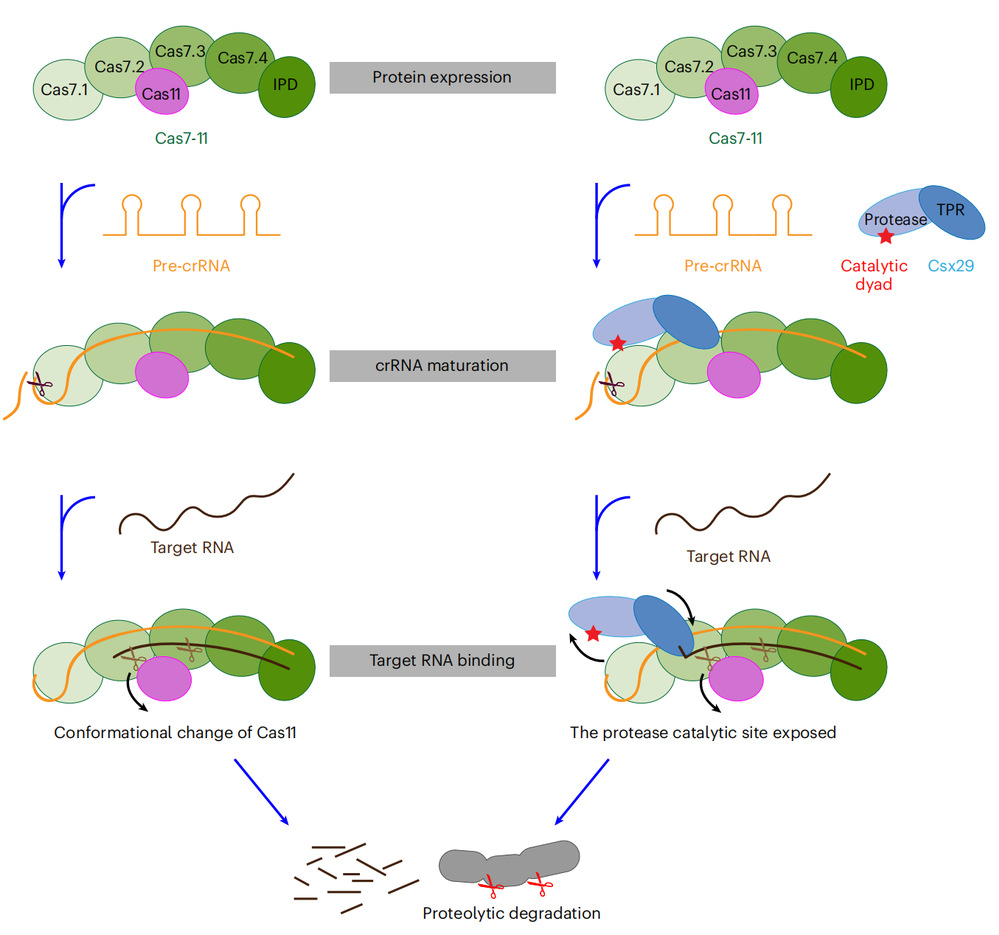
图5
天津医科大学张恒教授和武汉病毒所邓增钦研究员为论文的共同通讯作者。天津医科大学基础医学院于桂梅博士、研究生王枭燊、张祎和武汉病毒所博士生安启银为该论文共同第一作者。该研究得到了国家自然科学基金、中国科学院“百人计划”项目的支持。
参考文献:
1.Makarova, K. S. et al. Evolutionary classification of CRISPR–Cas systems: a burst of class 2 and derived variants. Nature Reviews Microbiology 18, 67-83 (2020).
2. Özcan, A. et al. Programmable RNA targeting with the single-protein CRISPR effector Cas7-11. Nature 597, 720-725 (2021).
3. van Beljouw, S. P. et al. The gRAMP CRISPR-Cas effector is an RNA endonuclease complexed with a caspase-like peptidase. Science 373, 1349-1353 (2021).
【作者介绍】

张恒博士,天津医科大学基础医学院教授博导。2020年9月加入天津医科大学建立结构生物化学实验室。课题组主要运用生物化学、结构生物学、微生物学和生物信息学等方法,研究微生物-宿主互作的分子机制,并开发基因编辑和检测工具。课题组自2020年9月份成立以来,在Nature Microbiology、Nature Chemical Biology、Nucleic Acids Research等杂志发表文章数篇(详见课题组网站www.zhanglab.life)。
热忱欢迎各位老师、同学合作交流!

